Komplementarlıq (molekulyar biologiya)
Komplementarlıq (molekulyar biologiya) — Molekulyar biologiyada, komplementarlıq hər biri "kilid və açar" prinsipinə tabe olan iki struktur arasındakı əlaqəni təsvir edir.[1] Təbiətdə, komplementarlıq DNT-nin təkrarlanması və transkripsiyasının əsas prinsipidir, çünki iki DNT və ya RNT ardıcıllığı arasında bölüşdürülən bir xüsusiyyətdir, beləliklə bir-birlərinə paralel olaraq nizamlandıqları zaman ardıcıllıqlardakı hər mövqedəki nükleotid əsasları komplementar olacaqdır.[2] Güzgüyə baxıb, əksini görmək kimi. Bu tamamlayıcı əsas cüt hüceyrələrə məlumatları bir nəsildən digərinə köçürməyə və hətta ardıcıllıqla saxlanılan məlumatların zədələnmələrini tapmaq və düzəltməyə imkan verir.[3]
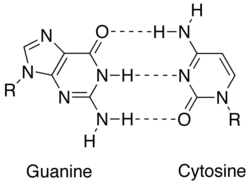

Komplementarlıq azotlu əsaslar arasındakı aydın qarşılıqlı təsirlər nəticəsində əldə edilir: adenin, timin (RNT-də urasil), guanin və sitosin. Adenin və guanin purinlər, timin, sitosin və urasil isə pirimidindir.[4] Purinlər pirimidinlərdən daha böyükdür. Hər iki növ molekul bir-birini tamamlayır və yalnız əks növ azot bazaları ilə baza cütləri yarada bilər.[5]
Nuklein turşuda nuklein əsaslar yalnız adenin ilə timin arasında və quanin ilə sitosin arasında təsirli olan hidrogen bağları ilə bir-birinə bağlanır. A = T baza tamamlayıcıda iki hidrogen, Q≡C baza cütdə isə üç hidrogen rabitə var.[6] Azotlu əsaslar arasındakı bütün digər konfiqurasiyalar ikiqat zəncir əmələ gəlməsinin qarşısını alacaqdır. DNT zəncirləri əks istiqamətlərə yönəldilmişdir, antiparallel sayılırlar.
| Nuklein turşuları | Nukleotid | Kompilementarlıq |
| DNT | adenin(A), timin(T), quanin(Q), sitozin(S) | A=T, Q≡S |
| RNT | adenin(A), urasil(U), quanin(Q), sitozin(S) | A=U, Q≡S |
DNT və ya RNT-nin tamamlayıcı zolağı azotlu əsasların komplementarlığına əsaslanaraq dizayn edilə bilər. Hər bir cüt, Q≡S ilə müqayisədə A = T, təxminən eyni məkanı tutur və bu da heç bir məkan təhrif etmədən bükülmüş DNT cüt zəncirinin əmələ gəlməsinə imkan verir. Azotlu əsaslar arasındakı hidrogen əlaqəsi də DNA cüt zəncirini sabitləşdirir.[7] Oxuma nəzarəti kimi DNT təmir mexanizmləri bir-birini tamamlamağa əsaslanır və uyğun olmayan nükleobazları çıxarıb DNT replikasiyası zamanı səhvləri düzəldə bilər. Ümumiyyətlə, bir DNT zəncirindəki zədələnmə, uyğunsuzluq təmirində, nükleotid eksizyonal təmirdə və baza eksizyonel təmirdə olduğu kimi, zədələnmiş bölgəni çıxarıb digər teldən məlumat kopyalamaq üçün tamamlayıcılıq istifadə edərək dəyişdirilə bilər.[8]
Tənzimləmə funksiyaları
redaktəTamamlayıcılıq nüklein turşunun qısa uzanması ilə kodlaşdırma bölgəsi və ya genin transkripsiyası nəticəsində baza cütləşməsinə səbəb ola bilər. Bu qısa nüklein turşu ardıcıllığı təbiətdə çox rast gəlinir və genləri susdurmaq kimi tənzimləyici funksiyaları yerinə yetirir.[9]
Bioinformatika
redaktəKomplementarlıq, DNT və ya RNT-də olan məlumatların tək bir zolaqda saxlanmasına imkan verir. Komplementarlıq iplik cDNT kitabxanalarında olduğu kimi şablondan və əksinə müəyyən edilə bilər. Eyni zamanda iki fərqli növün ardıcıllığını müqayisə etmək kimi analizlərə imkan verir. Qısaltmalar uyğunsuzluqlar (qeyri-müəyyənlik kodları) mövcudluğunda ardıcıllıqlar yazmaq və ya tamamlayıcıda (ambiqramda) əks ardıcıllığın oxunmasını sürətləndirmək üçün hazırlanmışdır.[10]
c-DNT kitabxanası
redaktəBir c-DNT kitabxanası, gen müəyyənləşdirmə və klonlama proseslərində faydalı bir istinad vasitəsi sayılan ifadə edilmiş DNT genlərinin toplusudur. C-DNT kitabxanaları, mRNT şablonunu DNT-yə köçürən əks transkriptaz (RT) RNT-yə bağlı DNT polimerazından istifadə edərək m-RNT-dan qurulur. Bu səbəbdən bir c- DNT kitabxanasında yalnız m-RNT-ya köçürülməsi nəzərdə tutulan əlavələr ola bilər. Bu proses DNT / RNT tamamlayıcılıq prinsipinə əsaslanır. Kitabxanaların son məhsulu, plazmidlərə yerləşdirilə bilən cüt telli DNT-dir. Beləliklə, cDNT kitabxanaları müasir tədqiqatlarda güclü bir vasitədir.[11]
Qeyri-müəyyənlik kodları
redaktəÜç nükleotiddən birini xüsusi olaraq istisna edən bir IUPAC kodu tamamlayıcı nükleotidi istisna edən bir IUPAC kodunu tamamlayıcı ola bilər. Məsələn, V (A, S və ya Q — "T" deyil) B ilə tamamlayıcı ola bilər (A, Q və ya T — "A deyil").[12]
Ambiqram
redaktəXüsusi simvollar tamamlayıcı əsaslar üçün uyğun (ambiqrafik) nüklein turşu notası yaratmaq üçün istifadə edilə bilər (məsələn, guanin = b, sitosin = q, adenin = n və timin = u), bütün DNT ardıcıllığını tamamlamağı asanlaşdırır. Məsələn, əvvəlki əlifba ilə buqn (QTSA) tərs çevrildikdə ubnq (TQAS, geri tamamlayıcı) oxuyur. Ambiqrafik təyinatlar palindromik ardıcıllıqlar kimi nuklein turşularının tamamlayıcı bölgələrini asanlıqla vizuallaşdırır. Bu xüsusiyyət, adi ASCII və ya hətta Unicode simvollarından daha çox xüsusi şriftlər və ya simvollar istifadə edilərkən artır.
İstinadlar
redaktə- ↑ Watson, James, Cold Spring Harbor Laboratory, Tania A. Baker, Massachusetts Institute of Technology, Stephen P. Bell, Massachusetts Institute of Technology, Alexander Gann, Cold Spring Harbor Laboratory, Michael Levine, University of California, Berkeley, Richard Losik, Harvard University ; with Stephen C. Harrison, Harvard Medical. Molecular biology of the gene (Seventh). Boston: Benjamin-Cummings Publishing Company. 2014. ISBN 978-0-32176243-6.
- ↑ Pray, Leslie. "Discovery of DNA structure and function: Watson and Crick". Nature Education. 1 (1). 2008: 100. 17 August 2022 tarixində arxivləşdirilib. İstifadə tarixi: 27 November 2013.
- ↑ Shankar, A; Jagota, A; Mittal, J. "DNA base dimers are stabilized by hydrogen-bonding interactions including non-Watson-Crick pairing near graphite surfaces". The Journal of Physical Chemistry B. 116 (40). Oct 11, 2012: 12088–94. doi:10.1021/jp304260t. PMID 22967176.
- ↑ Hood, L; Galas, D. "The digital code of DNA". Nature. 421 (6921). Jan 23, 2003: 444–8. Bibcode:2003Natur.421..444H. doi:10.1038/nature01410. PMID 12540920.
- ↑ Rawson JMO, Nikolaitchik OA, Keele BF, Pathak VK, Hu WS. Recombination is required for efficient HIV-1 replication and the maintenance of viral genome integrity. Nucleic Acids Res. 2018;46(20):10535–10545. DOI:10.1093/nar/gky910 PMID:30307534
- ↑ He, Y; Vogelstein, B; Velculescu, VE; Papadopoulos, N; Kinzler, KW. "The antisense transcriptomes of human cells". Science. 322 (5909). Dec 19, 2008: 1855–7. Bibcode:2008Sci...322.1855H. doi:10.1126/science.1163853. PMC 2824178. PMID 19056939.
- ↑ Katayama, S; Tomaru, Y; Kasukawa, T; Waki, K; Nakanishi, M; Nakamura, M; Nishida, H; Yap, CC; Suzuki, M; Kawai, J; Suzuki, H; Carninci, P; Hayashizaki, Y; Wells, C; Frith, M; Ravasi, T; Pang, KC; Hallinan, J; Mattick, J; Hume, DA; Lipovich, L; Batalov, S; Engström, PG; Mizuno, Y; Faghihi, MA; Sandelin, A; Chalk, AM; Mottagui-Tabar, S; Liang, Z; Lenhard, B; Wahlestedt, C; RIKEN Genome Exploration Research Group; Genome Science Group (Genome Network Project Core Group); FANTOM Consortium. "Antisense transcription in the mammalian transcriptome". Science. 309 (5740). Sep 2, 2005: 1564–6. Bibcode:2005Sci...309.1564R. doi:10.1126/science.1112009. PMID 16141073.
- ↑ Faghihi, MA; Zhang, M; Huang, J; Modarresi, F; Van der Brug, MP; Nalls, MA; Cookson, MR; St-Laurent G, 3rd; Wahlestedt, C. "Evidence for natural antisense transcript-mediated inhibition of microRNA function". Genome Biology. 11 (5). 2010: R56. doi:10.1186/gb-2010-11-5-r56. PMC 2898074. PMID 20507594.
- ↑ Kosaka, N; Yoshioka, Y; Hagiwara, K; Tominaga, N; Katsuda, T; Ochiya, T. "Trash or Treasure: extracellular microRNAs and cell-to-cell communication". Frontiers in Genetics. 4. Sep 5, 2013: 173. doi:10.3389/fgene.2013.00173. PMC 3763217. PMID 24046777.
- ↑ Reverse-complement tool Arxivləşdirilib 2022-04-16 at the Wayback Machine page with documented IUPAC code conversion, source code available.
- ↑ Rozak DA. "The practical and pedagogical advantages of an ambigraphic nucleic acid notation". Nucleosides Nucleotides Nucleic Acids. 25 (7). 2006: 807–13. doi:10.1080/15257770600726109. PMID 16898419.
- ↑ Rozak, DA; Rozak, AJ. "Simplicity, function, and legibility in an enhanced ambigraphic nucleic acid notation". BioTechniques. 44 (6). May 2008: 811–3. doi:10.2144/000112727. PMID 18476835.